个人简介:
读研时候使用了上百种生信软件/数据库,主要工作就是评估生信软件,靠比比软件就发了paper毕业了。
本期评测软件:esATAC;
esATAC: an easy-to-use systematic pipeline for ATAC-seq data analysis. ;
发表期刊:Bioinformatics; 发表时间: 2018年; 课题组: 清华汪小我课题组
论文地址:https://academic.oup.com/bioinformatics/article/34/15/2664/4924216
R包链接:https://bioconductor.org/packages/release/bioc/html/esATAC.html
Github源码:https://bioconductor.org/packages/release/bioc/html/esATAC.html
软件简介: 一套在win/linux/mac都能运行的ATAC-seq数据处理pipeline的R包,从fastq文件完成所有分析步骤,生成所有结果文件和可视化分析结果。
满分5分
用户体验:4分。
该软件是R包,R版本需求3.5以上,如果用户是3.4及以前的旧版本R必须升级,否则出错。但是R升级会导致老版本所装R包“丢失”,基本需要重装,这是R本身的缺陷。esATAC依赖包较多,还好大部分在都在bioconductor上,剩下几个手动装下,Java没有的也手动装下即可(jdk和jre免费下载地址)。R软件对版本环境硬性需求也是所有R软件的通病。很多几个版本未更新的R软件,不去修改源码都无法安装或者无法正常使用。
安装:我首先新建了一个R版本3.5.1的conda环境,装了Java然后进入R,执行标准安装命令。
source("https://bioconductor.org/biocLite.R")
biocLite("esATAC")
报错了根据提示基本可以解决,可以同时下载所需基因组等文件。需要的包比较多,折腾几下基本一天内可以搞定该软件的安装。
当然,相对于wdl、snakemake等pipeline语言,R对于新手以及R忠实铁粉而言会更容易上手。如若以后再conda cloud, docker cloud有会更棒。以现在条件评分4分。
软件功能:3.5分。
软件流程功能示意图:
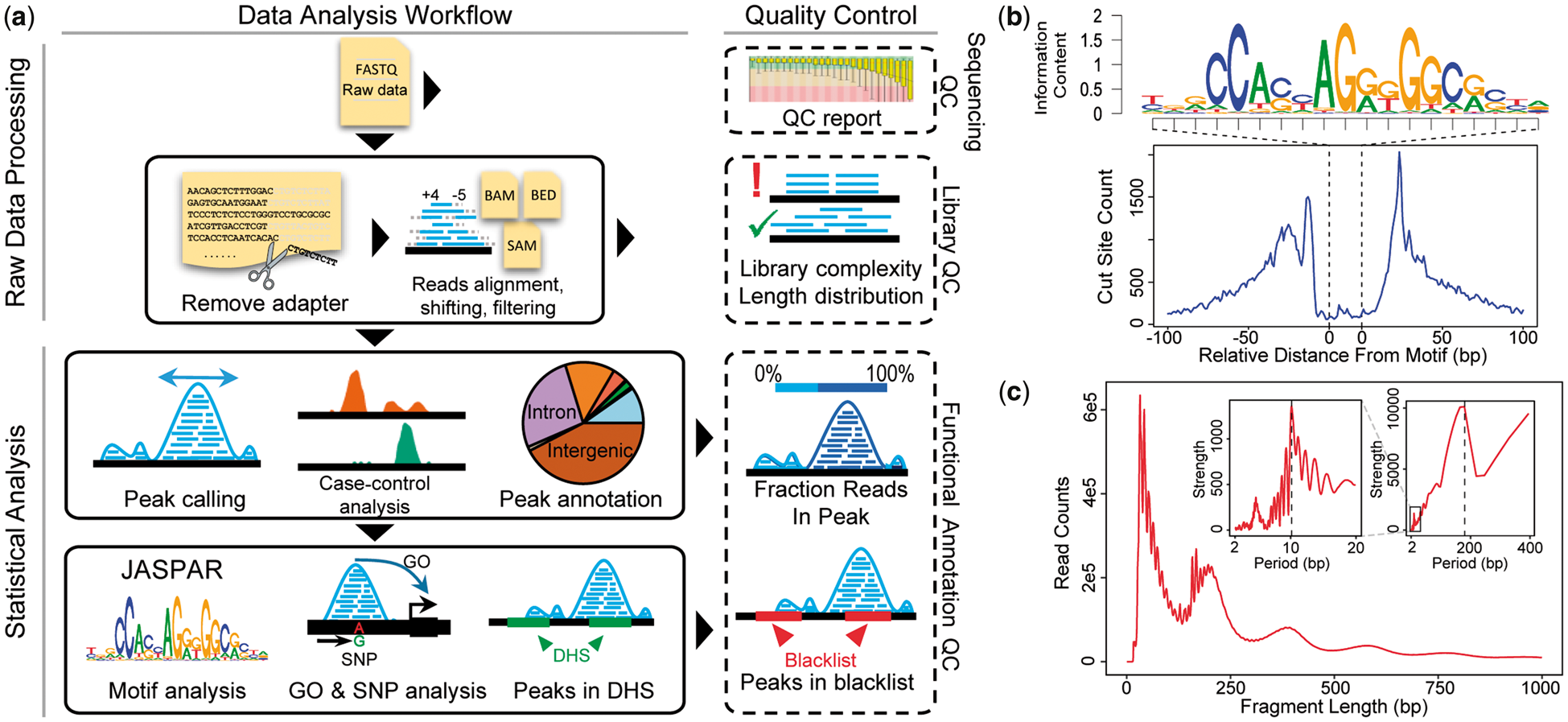
ATAC-seq分析质控后第一步就是mapping。esATAC的mapping用的是bowite2的R版本,同bowtie2一样消耗资源,额外还有R的开销,在一般个人主机上运行还是较慢。处理大量ATAC-seq数据时依旧需要服务器或者集群来实现,无法实现R即开即用快速分析的优势。esATAC的质控等功能类似chilin)的pipeline,但是性能上并未更优。
另一个核心功能call peak算法用的是F-seq。F-seq是年代久远、且比较适合新手的peak caller,可调参数很少,得到Peak很多,随便做都有结果。实际F-seq这个软件用来看全局信号还是很合适的。在过去几年个人对F-seq的评测工作中,其缺点是非常明显,且致命的。
- peak过多,太多noise。会得到常规软件10倍的peak,但是很多都非常短(几bp)。
- 大BAM文件会内存爆炸(JAVA的),500g以上内存比较保险。
- 给算法已经很老了,08年发的paper,而13年后就再没更新了。
相对于用MACS系列,HOMER系列或者其他更新的软件,该pipeline在此有较大瓶颈,也影响下游分析结果质量好坏。
下游分析。该软件下游分析还是比较全面的,包含一个atac的pipeline该有的分析项目,能够实现主要科研问题的需求。
R自身瓶颈加上F-seq软件缺陷,因此评分3.5分
领域创新:4分。
第一个公开发表的R的专门用于ATAC-seq的pipeline,并且能够在R体系中整合上下游的所有流程,还是挺难能可贵的。
其他大佬的ATAC的pipeline,谷歌搜下就跳出来了,conda、docker、集群、amazon、本地都可以快速安装,其中参数的调优和配置水平还是很高的,只是没发paper罢了。因此,esATAC软件从软件或者pipeline的角度,其创新度并不算非常突出。因此评分4分。
小结:
作为国人新出的R包版ATAC-seq pipeline,的确为很多R用户和ATAC-seq研究者提供了很大的便利。虽然有些小局限,仍不失国产软件良心之作~
沙湿兄评软件持续更新中~ 欢迎大家拍砖指正错误~ 也欢迎大家提出生信新软件/数据库评测需求~
附:个人软件比较工作