2. 测序结果质量评估
2.1 测序错误率分布检查
每个碱基测序错误率是通过测序Phred数值(Phred score, Qphred)通过公式(公式1 :Qphred = -10log10(e))转化得到,而Phred 数值是在碱基识别(Base Calling)过程中通过一种概率模型计算得到,这种模型可以准确地预测碱基判别的错误率。Phred分值,不正确的碱基识别率,碱基正确识别率以及Q-score的对应关系如下表1所显示:
表1 质量与错误率对照
| 测序错误率(E) | 碱基正确识别率 | 测序质量值(Q) |
|---|---|---|
| 5% | 90% | 13 |
| 1% | 99% | 20 |
| 0.1% | 99.9% | 30 |
| 0.01% | 99.99% | 40(max Q) |
(1)测序错误率随着测序序列(Sequenced Reads)长度的增加而升高。这是由测序过程中化学试剂的消耗导致的,为Illumina高通量测序平台所具有的特征。
(2)前6个碱基具有较高的测序错误率,此长度恰好为RNA-seq建库过程中反转录所需的随机引物长度。前6个碱基测序错误率较高是因为随机引物和RNA模版的不完全结合(Jiang et al.)。
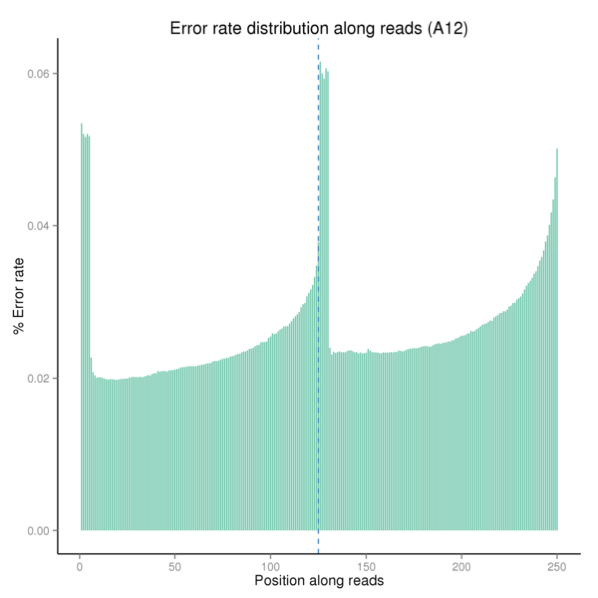
图3.2.1 测序错误率分布图
2.2 碱基含量分布检查
通过碱基分布示意图,图3.2.2,我们可以观测测序过程碱基均衡性,一般来说,因采用随机引物扩增但随机引物种类有限而导致reads的前10个碱基的比例不均衡,会出现波动是正常现象。在此波动之后的碱基互补配对原理GC及AT碱基对会分别均衡分布。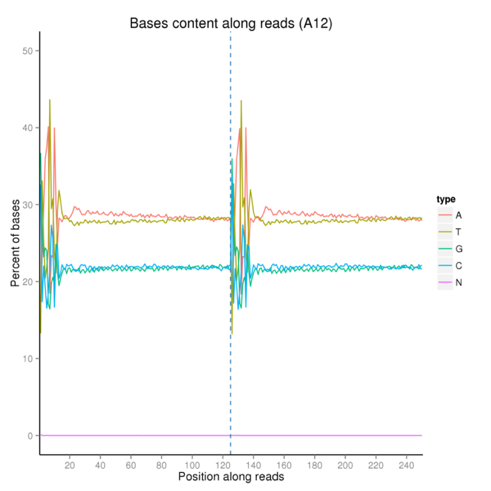
图3.2.2 碱基分布示意图
2.3 Adapter Content分析
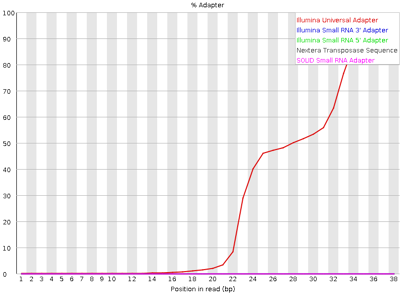
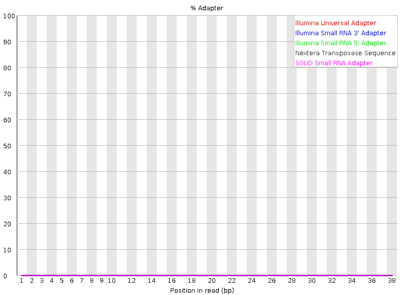
图3.2.3 接头去除前(左图)后(右图)Adapter Content统计图
注:1、Adapter Content反映序列reads在每个接头序列文库的立即百分比率;
2、正常情况,在完全去除接头情况下,序列reads在接头序列文库中占比是趋于零的,即Adapter Content图形中曲线贴近基线;
3、接头信息:3’adapter,5’-AGATCGGAAGAGCACACGTCT-3’;
5’adapter,5’-GTTCAGAGTTCTACAGTCCGACGATC-3;
4、这里只展示treat1样本去除接头前后的Adapter Content统计结果。
2.4 测序数据过滤
测序下机得到的原始序列,包含有带接头及低质量的reads,为了保证信息分析质量,必须对raw reads进行过滤,得到clean reads,后续分析都基于clean reads。数据的处理大致如下:(1) 去除带接头(adapter)的reads;
(2) 去除N(N表示无法确定碱基信息)的比例大于10%的reads;
(3) 去除低质量reads(Qphred <= 20 的碱基数占整个 read 长度的 50%以上的 reads)。
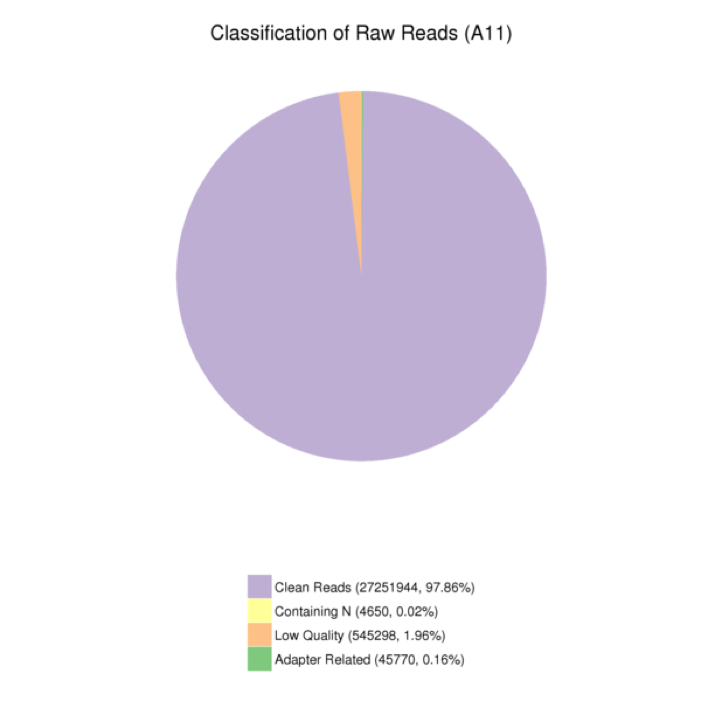
图3.2.4 原始数据组成
注:不同颜色的比例分别代表不同成分比例(1)Adapter related:因有接头,过滤掉的 reads数及其占总 raw reads数的比例。
(2)Containing N:因 N含量超过 10%,过滤掉的 reads数及其占总raw reads数的比例。
(3)Low quality:因低质量,过滤掉的reads数及其占总raw reads数的比例。
(4)Clean reads:最终得到的 clean reads 数及其占总 raw reads 数的比例。