2. lncRNA trans-regulation预测
lncRNA trans-regulation预测是基于由microRNA介导的miRNA-lncRNA的sponge原理,定位调控关键基因。
在lncRNA的众多功能中,lncRNA会通过结合microRNA来抑制microRNA对mRNA的抑制或降解的作用(这里,lncRNA被形象的比喻成海绵,被吸收的microRNA就是水滴)。我们在这里通过预测的方法预测潜在的lncRNA-microRNA-mRNA相互作用,从而定位关键lncRNA。
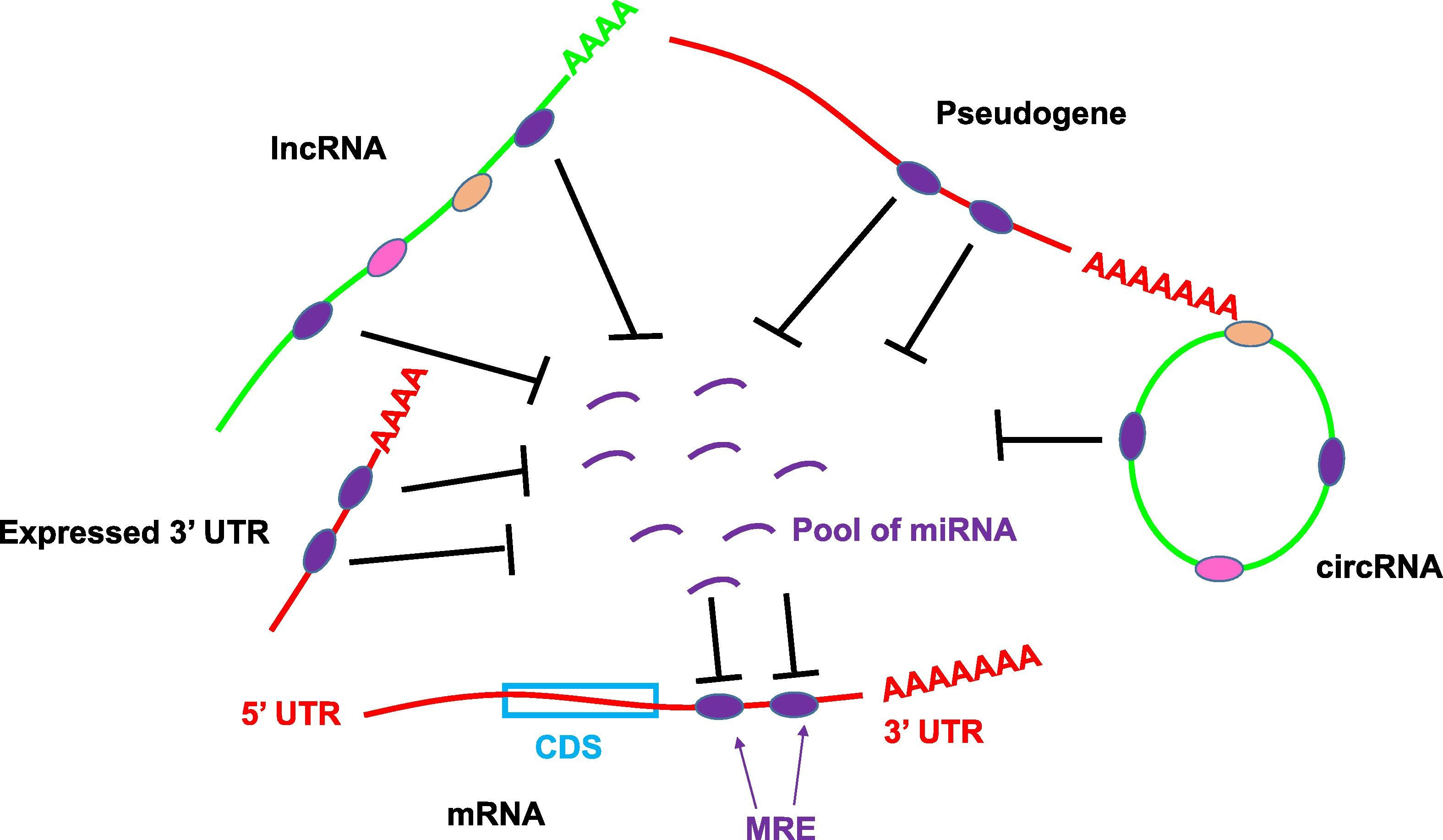
图5.2 lncRNA反式调控示意图
我们首先对每一个lncRNA通过计算机软件,计算潜在的组成“lncRNA-microRNA-mRNA匹配对”。进一步,考虑到基于这一海绵原理结合的lncRNA与mRNA会呈现正向表达的关系,所以我们再检测mRNA和lncRNA的基因表达量是否协调一致,从而定位高度正向共表达的“lncRNA-microRNA-mRNA匹配对”。在得到匹配对以后,我们可以通过mRNA的功能推测与其匹配的lncRNA功能。
老师可以通过自己倾向的几个原则之一,利用excel 对我们的输出结果自行进行简单操作,从而定位关键基因:
1)老师可以通过mRNA或者lncRNA的差异表达程度,进一步过滤筛选更为严格的“lncRNA-microRNA-mRNA匹配对”。(我们的输出文件会有每一个lncRNA以及mRNA的差异表达程度);
2)与老师感兴趣的mRNA有关联的lncRNA是关键lncRNA。如果老师有感兴趣的mRNA的话,通过excel搜索命令,就可以找到与之关联的关键lncRNA。
3)案例:发表于oncogene的文献A long noncoding RNA critically regulates Bcr-Abl-mediated cellular transformation by acting as a competitive endogenous RNA的作者就是通过该分析,定位了通过海绵作用介导的lncRNA-mRNA 搭配对,并进一步定位到了与PTEN(tumor suppressor)相关的关键lncRNA。
表8 lncRNA反式调控示意图
| lncRNA_name | lncRNA_logFC | ncRNA_p_value | mRNA_name | mRNA_logFC | mRNA_p_value | microRNA | TargetScan_Score_mRNA | TargetScan_Score_lncRNA |
|---|---|---|---|---|---|---|---|---|
| HOXB-AS1 | 0.69 | 0.03 | TKT | -0.78 | 0.04 | hsa-miR-485-5P,hsa… | -0.10,-0.33,… | -0.12,-0.17,… |
| RP11-443A13.5 | -0.58 | 0.03 | SLC27A1 | -0.8 | 0.03 | hsa-miR-124a,hsa… | -0.09,-0.08,… | -0.07,-0.07,… |
| HOXB-AS1 | 0.69 | 0.03 | SLC27A1 | -0.8 | 0.03 | hsa-miR-136,hsa… | -0.07,-0.06,… | -0.06,-0.12,… |
| RP11-498C9.13 | 0.53 | 0.04 | TKT | -0.78 | 0.04 | hsa-miR-520a,hsa… | -0.03,-0.03,.. | -0.10,-0.10,… |
| RP11-498C9.13 | 0.53 | 0.04 | CDC42SE1 | 0.63 | 0.02 | has-378,hsa… | -0.20.-0.30,.. | -0.13,-0.09,… |
注:
(1)第一列为lncRNA,第二列和第三列是这个lncRNA的差异表达log(fold-change)和显著性p-value;第四列为mRNA,第五列和第六列是这个mRNA的差异表达log(fold-change)和显著性p- value。第七列是介导sponge作用的microRNA列表(因为是用软件targetscan预测的,所以数量往往很多)。第八列和第九列分别是mRNA 和lncRNA 与每一个第七列中的microRNA 的结合强度;
(2) 文件结果汇总在lncRNA/trans-regulation中的inquiry_trans_regulation.xls文件中;
(3)对应文件夹中的lncRNA_targetScan_results_hg19和mRNA_targetScan_results_hg19是涉及到的microRNA与lncRNA,mRNA结合强度预测的相关参数。具体说明请参见targetscan官网http://www.targetscan.org;
(4)如果是windows系统,请用excel文件打开所有的文件。